训练AlphaFold2以及使用其进行蛋白质结构预测的推理计算,需要怎样的计算力支持?
来源:www.it778.com | 发布时间:2022年05月13日训练AlphaFold2以及使用AlphaFold2进行蛋白质结构预测的推理计算,需要怎样的计算力支持?济南戴尔服务器代理商告诉你,戴尔科技中国研究院以及戴尔数据中心业务部解决方案团队,通过在GitHub下载AlphaFold2模型代码,部署在戴尔Dell PowerEdge XE8545服务器上,使用NVIDIA A100 GPU测试AlphaFlod2对68-2750个氨基酸残基组成的不同大小的蛋白质进行3D结构预测,对AlphaFold2的计算性能和特性进行评估。

戴尔PowerEdge XE8545是戴尔科技最新推出的15G服务器家族中,专门针对AI GPU计算进行设计和优化的加速服务器。4U空间内可以支持4张A100 GPU加速卡,GPU之间通过NVLink实现600GB/s的pear-to-pear高速直连通信。
测试环境硬件及软件配置如下:
●AMD EPYC 7713 64-Core Processor × 2
●1024 GB memory
●Nvidia A100 GPUs × 4, 80GB/500W
●CentOS Linux 7.0
●Python 3.8.0, TensorFlow 2.5.0
●CUDA 11.5, cuDNN 8.3
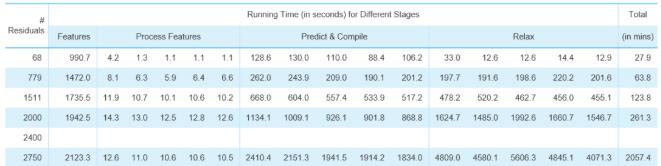
戴尔XE8545服务器推理68-2750个氨基酸残基组成的蛋白质的3D结构预测耗费的计算时间如下表所示(Top 1模型,即推荐置信度佳的模型),使用单张A100推理计算时间从19.3分钟到2个半小时不等。

如果按照DeepMind论文Top5模型,XE8545单卡A100推理计算时间如下:
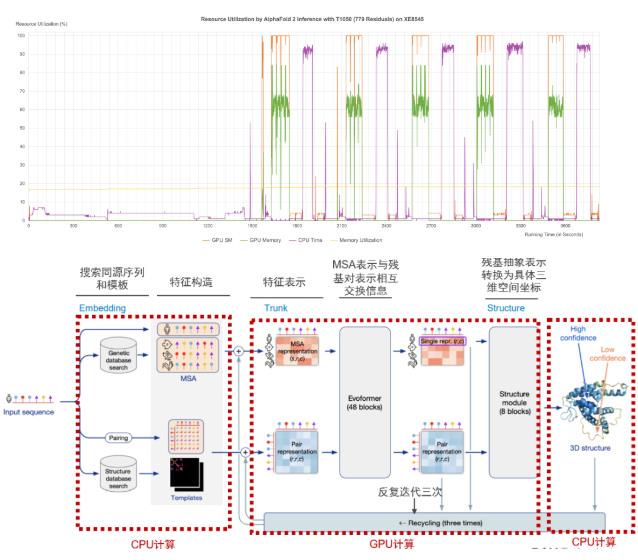
通过性能日志分析,我们可以明显地看到AlphaFold2在推理过程中,由CPU和GPU交替计算,第 一阶段同源序列搜索、模版搜索及特征构造,以及后阶段3D结构生成的计算过程主要由CPU计算;中间第二个阶段Evoformer神经网络和结构模块计算则主要由GPU进行计算。而戴尔XE8545服务器所提供的强劲GPU算力与AMD 多核CPU算力(128核),则能够确保AlphaFold2在规定时间内完成一个大型的蛋白质3D结构的预测计算。
我们也对比了不同GPU对于AlphaFold2推理计算性能的影响。我们选取了一台戴尔7750工作站,配置一张NVIDIA RTX5000显卡,对蛋白质结构预测(Top 1模型)计算性能进行对比,对比结果如下表所示:
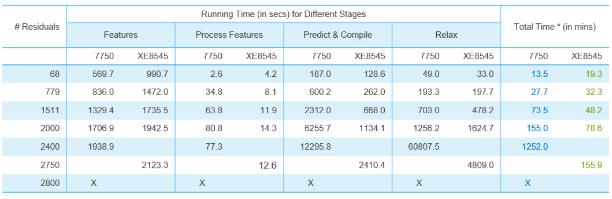
实验数据显示:当蛋白质规模很小的时候,企业级与消费级GPU性能相差不大;越大的蛋白质,使用A100结构预测加速性能越明显。预测1511个残基的蛋白质3D结构,戴尔XE8545服务器+A100耗费时间是RTX5000的65%;预测2000个残基的蛋白质3D结构,XE8545耗费的时间只有RTX5000的50%。
我们可以看到,当预测2800个残基的蛋白质结构时,RTX5000由于显存容量和算力的限制,无法完成结构预测工作,而XE8545仍然以小时级的时间顺利完成同等规模的蛋白质结构预测。
从模型训练的角度来看,Alphafold2以及后续出现的类似的蛋白质结构预测模型,由于采用Transformer机制,模型训练需要非常高的计算力,通常需要64-512张GPU组成计算集群,采用分布式训练机制,才能在比较短的时间内实现模型收敛。
DeepMind在论文中谈到,训练AlphaFold2模型使用128块Google TPU芯片,接近2周时间完成模型训练。2022年3月,上海交通大学与潞晨科技发布的FastFold模型,使用256张A100 GPU进行初始训练和512张A100进行Fine-tuning,2.81天完成模型训练。
戴尔科技AI GPU分布式训练解决方案,能够提供高速GPU计算、小文件IO快速读写(蛋白质数据库存在大量小文件)和高带宽低延迟地网络通信,帮助用户实现在深度学习框架下分布式训练的自动化实现与性能优化,轻松应对AI时代浪潮。
【相关文章】
【相关产品】
 微信号:扫码加微信
微信号:扫码加微信








 添加微信
添加微信
 联系我们
联系我们
 电话咨询
电话咨询